抽象工厂模式
简言之,就是业务逻辑需要创造一套套对应的组件。在不同条件下,需要的配套组件不同,但是这些组件都执行相似的功能。调用者直接接收一个工厂类,这个工厂类对应了一系列组件。不同的工厂制造不同的组件,但都遵循相同的接口。
换句话说,工厂实际上是对产品的一种打包!抽象工厂提供了对工厂的多态支持。
123456789101112131415161718192021222324252627282930313233343536373839404142434445464748495051525354555657585960616263646566676869707172737475767778798081828384858687888990919293949596979899100101102103104105106107108class GUIFactory { public: virtual Button* createButton() const = 0; virtual Checkbox* createCheckbox() const = 0;};class ...
3DSlicer 笔记
Segmentation
generate from seed 很好用
Nuclear Medicine 药物与显像技术
放射性药物
体外放射分析用试剂盒不属于放射性药物,而应归类为试剂
基本特性
放射性
体内效应取决于被标记物特性
脱标与辐射自分解
radiotion self-decomposition 某些被标记物对射线作用敏感,射线作用下可发生化学结构变化或生物活性消失,导致放射性药物体内生物学行为改变
计量单位:放射性活度,而非化学量(物质的量)
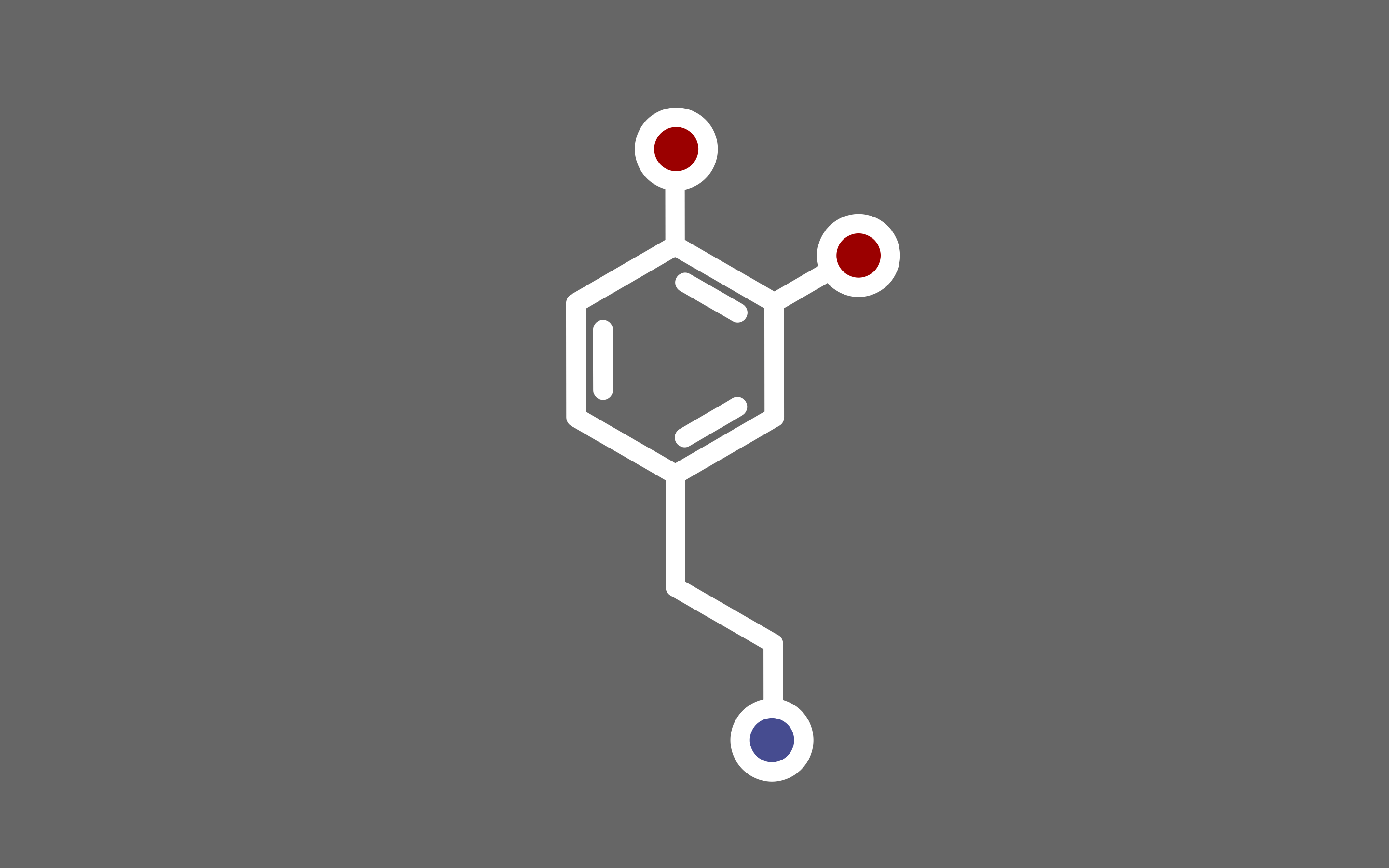
99mTc\rm {}^{99m}Tc99mTc标记,一次370MBq(10mCi),化学量仅e-10~e-9mol
有效使用期
诊断用放射性药物
衰变方式:
99mTc\rm {}^{99m}Tc99mTc:同质异能衰变,γ
201Tl,111In,67Ga,123I\rm {}^{201}Tl, {}^{111}In, {}^{67}Ga, {}^{123}I201Tl,111In,67Ga,123I:电子俘获衰变,发射特征X线或γ
11C,15O,13N,18F\rm {}^{11}C, {}^{15}O, {}^{13}N, {}^{18}F11C,15O,13N,18F:β+\beta^+β+
带电荷射线电离能力强,容易对 ...
Nuclear Medicine 物理基础与仪器
物理基础
同位素、核素、同质异能素
核素的表示
ZAXN\rm {}^A_ZX_N
ZAXN
其中A为质量数,Z为质子数,N为中子数。
激发态的原子核用m表示:
99mTc\rm {}^{99m}Tc
99mTc
衰变
α衰变,空气中只能穿透几厘米
β衰变,分β+\beta^+β+和β−\beta^-β−,其中后者是与电子碰撞产生湮灭辐射,转变为两个能量同为511keV,方向相反的γ光子
γ衰变,ZAmX→ZAY+γ\rm {}^{Am}_ZX \rightarrow {}^A_ZY+\gammaZAmX→ZAY+γ,同质异能跃迁isomeric transition IT
电子俘获:原子核俘获一个轨道电子,导致一个质子转变为中子,释放一个中微子(类似正电子衰变)
特征X射线:外层电子补充被俘获电子空缺轨道,直接释放能量
俄歇电子auger electron:释放的能量传递给更外层电子,使其释出
γ辐射:原子核处在激发态,直接释放能量
内转换电子:原子核能量传递给轨道电子,使其释出
衰变速度
dNdt=−λN\frac{dN}{dt} = -\lambda N
dtd ...
生存分析笔记
https://lifelines.readthedocs.io/en/latest/Survival analysis with lifelines.html
前置知识
(非)参数估计:“参数” 通常指的是用来描述数据集分布特征的量,例如均值、方差、比例或回归系数
参数估计:指基于样本数据估计总体参数(如总体均值或方差)的过程,通常假设数据遵循某种已知的概率分布(如正态分布)
非参数估计:不依赖于数据遵循特定的分布假设
参数估计有一个“拟合”过程,非参数则没有
123456789from lifelines import WeibullFitterT = data['T']E = data['E']wf = WeibullFitter().fit(T, E)wf.print_summary()wf.plot_cumulative_hazard()
比较分布手段
QQplot:将它们的分位数(quantiles)进行对比。它通常用于检验一个数据集是否遵循某个特定分布,例如正态分布
X轴表示理论分布的分位数,而Y轴表示样本数据 ...
PET图像重建关键性参数笔记
template_sinogram.hs文件用于配置重建参数。
1234567891011121314151617181920212223242526272829303132333435363738394041424344454647!INTERFILE :=!imaging modality := PTname of data file := template_sinogram.hsoriginating system := userdefined!version of keys := STIR3.0!GENERAL DATA :=!GENERAL IMAGE DATA :=!type of data := PETimagedata byte order := LITTLEENDIAN!PET STUDY (General) :=!PET data type := Emissionapplied corrections := {None}!number format := float!number of bytes per pixel := 4number of ...
树莓派4B配置OpenWRT记录
步骤记录
rpi的物理网口作为wan口,无线网络作为lan
12flowchart LR 光猫 --> 主路由 -->|以太网| rpi -->|无线| 上网设备
刷入immortalwrt
有线网连接树莓派,可能需要手动配置一下ip地址,使设备和树莓派处在同一个网段中
将lan口配置为:
协议:静态地址
设备:br-lan 设备:phy0-ap0
ipv4地址:192.168.8.1(可以选一个和主路由不冲突的)
ipv4网关留空(会默认配置为192.168.31.1,也就是主路由的网关)
新建一个wan口:
协议:DHCP客户端
设备:br-lan 设备:eth0
配置防火墙:在网络-防火墙-区域中,设置lan到wan的规则中,wan口启用IP动态伪装。这个也称为源NAT/IP masquerading
解释
想进行代理有大概两种思路:
把rpi视作一个上网设备,配置设备为br-lan,协议静态地址,ipv4地址选一个与主路由相同网段的(例如192.168.31.150之类的),并在DHCP里勾选忽略此接口(也就是此接口不提供DHCP服务, ...
Personalized Treatment Planning for Radiopharmaceutical Therapy
Personalized Treatment Planning for Radiopharmaceutical Therapy
Pre-therapy imaging (PET/CT)
Dose-estimation: Intensively investigated and partially solved (planar / SPECT/CT)
Treatment
Dose prediction
Dose effect
Treatment outcome
Adverse events
Vitalize the virtual patient
Physiologically-based pharmacokinetic (PBPK) model
Simulate time-course of radioligand uptake in organs of virtual patient
Organs & tumor: homogenous
Simulate PET imaging using realistic PET simulator
Dose calc ...
SPM学习笔记
开启SPM12
1spm pet
基本操作
12345I = dicomread(dicomFile)imshow(I, 'DisplayRange', []) % 显示DICOM图像info = dicominfo(dicomFile) % 显示元信息% info.AcquisitionTime较重要
计算SUV:使用ImCalc输入计算公式后,Display选中计算结果
图像预处理
Spatial Registration, Normalization
Registration: Rigid Transformations (rotations, translations…)
Normalization: Transform to Montreal Neurology Institute (MNI) Template
Smoothing: e.g. Gaussian kernel, improve SNR, minimize differences between normalized images, increase validity of statist ...